-
Notifications
You must be signed in to change notification settings - Fork 27
Commit
This commit does not belong to any branch on this repository, and may belong to a fork outside of the repository.
- Loading branch information
Showing
2 changed files
with
179 additions
and
0 deletions.
There are no files selected for viewing
This file contains bidirectional Unicode text that may be interpreted or compiled differently than what appears below. To review, open the file in an editor that reveals hidden Unicode characters.
Learn more about bidirectional Unicode characters
This file contains bidirectional Unicode text that may be interpreted or compiled differently than what appears below. To review, open the file in an editor that reveals hidden Unicode characters.
Learn more about bidirectional Unicode characters
| Original file line number | Diff line number | Diff line change |
|---|---|---|
| @@ -0,0 +1,178 @@ | ||
| --- | ||
| date: 2024-07-14 | ||
| comments: true | ||
| --- | ||
| # 生信爱好者周刊(第 131 期):2023诺贝尔生理医学奖给了mRNA疫苗技术,没有终身教职的她拿奖了 | ||
|
|
||
| 这里记录每周值得分享的生信相关内容,周日发布。 | ||
|
|
||
| 本杂志开源(GitHub: [openbiox/weekly](https://github.com/openbiox/weekly)),欢迎提交 issue,投稿或推荐生信相关内容。 | ||
|
|
||
| [「生信周刊讨论区(语雀)」](https://www.yuque.com/shixiangwang/bioinfo "「生信周刊讨论区(语雀)」") | [「生信讨论区(Gitter)」](https://gitter.im/ShixiangWang/community "「生信讨论区(Gitter)」") | ||
|
|
||
| ## 封面图 | ||
|
|
||
|  | ||
|
|
||
| ## 本周话题:[2023诺贝尔生理医学奖给了mRNA疫苗技术,没有终身教职的她拿奖了 ](https://mp.weixin.qq.com/s/r9DRuhB1ISaMcUX-BEw-2Q) | ||
|
|
||
|  | ||
|
|
||
| >2023年诺贝尔生理学或医学奖宣布授予德国生物技术公司BioNTech的卡塔琳·卡里科(Katalin Karikó)、美国宾夕法尼亚大学教授德鲁·魏斯曼(Drew Weissman),以表彰他们在核苷碱基修饰方面的发现,这些发现使得针对 COVID-19 的有效 mRNA 疫苗得以开发。 | ||
| > | ||
| >两位科学家的突破研究使得高效率的新冠mRNA疫苗得以被快速研发,也已凭此获得了多项大奖,包括2022年美国科学突破奖(Breakthrough Prize)、2021年拉斯克基础医学研究奖(The Lasker Awards)、2023年盖尔德纳奖等。 | ||
| ## 生信研究 | ||
| 1. [Nucleic Acid Research | 北京基因组所(国家生物信息中心)发布空间转录组综合资源存储库CROST](https://mp.weixin.qq.com/s/qLJzYy4ZZCw2aiwg79nljA) | ||
|
|
||
|  | ||
|
|
||
| 空间转录组学是生命科学领域的重大突破,而CROST数据库的推出无疑是该领域的里程碑事件。这个由中国科学院北京基因组研究所主导开发的综合资源存储库,整合了来自8个物种、35种组织和56种疾病的1000多个高质量空间转录组数据集,并提供了全面的生物信息分析。CROST不仅方便研究人员访问海量数据,还提供交互式可视化分析环境和一站式分析平台,即使没有编程基础的用户也能轻松开展研究。它尤其有助于临床医生快速评估特定癌症基因的表达、甲基化和拷贝数变异情况,推动精准医疗发展。 | ||
|
|
||
| - 论文链接:https://doi.org/10.1093/nar/gkad782 | ||
|
|
||
|
|
||
| 2. [Nature | Olink Explore里程碑研究开启大规模人群蛋白质组学新时代](https://mp.weixin.qq.com/s/AYxQ8zlXoLPpoGbCUwY6eg) | ||
|
|
||
|  | ||
|
|
||
| 瑞典Olink公司在《自然》杂志发表的三篇研究,利用Olink Explore平台在54,000名参与者样本中分析了3,000种蛋白标志物,揭示了14,000多个新的遗传关联,推动了大规模人群蛋白质组学研究。这些发现有助于识别生物途径,加速药物开发,促进精准医学的发展。Olink的技术平台因其高特异性和精确度,为多组学整合和生物标志物发现提供了重要工具。 | ||
|
|
||
| - 论文链接: | ||
| 1. Sun et al. (2023) Plasma proteomic associations with genetics and health in the UK Biobank. Nature, DOI: 10.1038/s41586-023-06592-6 | ||
| 2. Dhindsa et al. (2023) Rare variant associations with plasma protein levels in the UK Biobank. Nature, DOI: 10.1038/s41586-023-06547-x | ||
| 3. Eldjarnet al. (2023) Large-scale plasma proteomics comparisons through genetics and disease associations. Nature, DOI: 10.1038/s41586-023-06563-x | ||
|
|
||
|
|
||
| 3. [Signal Transduction and Targeted Therapy丨这项研究为EBV阳性胃癌患者提供新的治疗方向](https://mp.weixin.qq.com/s/VUF9r-LdVqylOiKBNJgCWw) | ||
|  | ||
|
|
||
| 中山大学肿瘤防治中心徐瑞华教授团队在国际知名期刊STTT上发表重要研究成果,该研究团队通过动态单细胞测序技术,研究人员精细解析了EBV阳性和阴性胃癌免疫微环境的差异,发现EBV阳性胃癌具有更丰富的T、B细胞浸润,特别是ISG-15+CD8+T细胞亚群可预测更好的免疫治疗响应。研究还发现,LAG-3抑制剂可能是EBV阳性胃癌患者的潜在新靶点,这一发现已在临床试验中初步验证。该研究从细胞和分子水平阐明了EBV感染影响胃癌免疫治疗疗效的机制。 | ||
|
|
||
| - 论文链接:https://doi.org/10.1038/s41392-023-01622-1 | ||
|
|
||
| 4. [Nature Biotechnology | 基于分层图像特征提取的AI辅助新工具,可以超分辨率预测空间基因表达,实现更快速的癌症病理诊断](https://mp.weixin.qq.com/s/hjQ43i_PdCGW3xbaGRhtFA) | ||
|  | ||
|
|
||
| 宾夕法尼亚大学李明瑶教授团队开发了一种名为iStar的AI辅助工具,可以超分辨率预测空间基因表达,实现更快速精准的癌症病理诊断,并发表在Nature Biotechnology上。iStar采用分层图像特征提取方法,捕获组织学图像的局部细胞结构和全局组织结构信息,并结合空间转录组学数据训练神经网络模型,以接近单细胞的分辨率预测未观察到的超分辨率基因表达。此外,iStar在多种肿瘤类型中展现出优异表现,能自动检测出肉眼难以识别的肿瘤细胞,有望辅助确定癌症手术切除范围。最后,iStar的计算速度比其他AI工具快200多倍。 | ||
|
|
||
| - 论文链接:https://www.nature.com/articles/s41587-023-02019-9 | ||
| ## 博文资讯 | ||
|
|
||
| 5. [如何制作CPU-基于图片的简单解释](https://blog.robertelder.org/how-to-make-a-cpu/) | ||
|
|
||
|  | ||
|
|
||
| 本文通过图文结合介绍计算芯片如何从原始材料中被制作出来。 | ||
|
|
||
| 6. [Promise: 异步编程的强大工具](https://mp.weixin.qq.com/s/atFlcMdulXcaC1dZidOW0Q) | ||
|
|
||
|  | ||
|
|
||
| 这篇文章讲述了JavaScript中的Promise对象,它用于简化异步编程。Promise有三种状态:Pending、Fulfilled和Rejected。文章解释了如何使用Promise进行链式调用和错误处理,并提供了一些最佳实践,强调了Promise在提高异步代码可维护性方面的重要性。 | ||
|
|
||
|
|
||
|
|
||
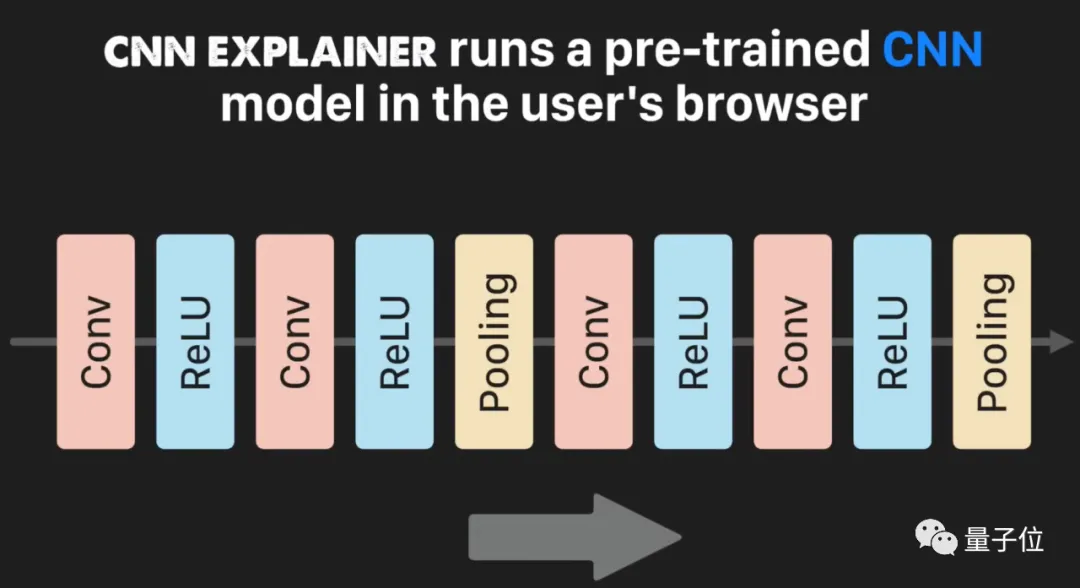
| 7. [一位博士把整个 CNN 都给可视化了,可交互有细节,每次卷积 ReLU 池化都清清楚楚](https://mp.weixin.qq.com/s/pCH1oaNOViJekw7nh3QOGQ) | ||
|
|
||
|
|
||
|  | ||
|
|
||
|
|
||
| CNN是什么?美国有线电视新闻网吗? | ||
|
|
||
| 每一个对AI抱有憧憬的小白,在开始的时候都会遇到CNN(卷积神经网络)这个词。但每次,当小白们想了解CNN到底是怎么回事,为什么就能聪明的识别人脸、听辨声音的时候,就懵了,只好理解为玄学。 | ||
|
|
||
| 好吧,维基百科解决不了的问题,有人给解决了。 | ||
|
|
||
| 这个名叫CNN解释器在线交互可视化工具,把CNN拆开了揉碎了,告诉小白们CNN究竟是怎么一回事,为什么可以辨识物品。 | ||
|
|
||
| 8. [Python软件包管理的最佳实践](https://mp.weixin.qq.com/s/8M5ssxmGXYECrAwpACoxgQ) | ||
|
|
||
|
|
||
|  | ||
|
|
||
|
|
||
| 今天,很偶然地,当我觉得不再需要某个包时,终于祭出了 pip uninstall 命令。本以为它能够依然智能地查出那些曾经搭便车被安装到系统中的包,并加以清理。然而,没想到,它只删除了我所指定的那个包,全然忘记了其他那些依赖项的存在。 | ||
|
|
||
| 经翻查文档和询问ChatGPT,才发现人家 pip 就是这么设计的,管装不管卸,只要一经安装到系统,就“一视同仁”对待,并不区分是否来自搭便车。这可真要命!原来从我第一次运行 pip install XXX 起,Python环境就已无法保持整洁了…… | ||
|
|
||
| 所幸,会为这个问题烦恼的,并非我一个人。为此,有不止一个包,能够帮助解决这个问题。这些包大同小异,原理也差不多,这里就选择其中一个,pip-autoremove,来加以介绍。 | ||
|
|
||
|
|
||
| ## 工具 | ||
|
|
||
| 9. [Python 库,用 Gooey 为你的程序添加GUI](https://mp.weixin.qq.com/s/0dIRzA4pJiuE_3CymxnIGA) | ||
|
|
||
|
|
||
|  | ||
|
|
||
|
|
||
| 大家有没有遇到过这样的情况:你写了一个非常棒的命令行程序,但当你分享给朋友或同事时,他们却因为害怕命令行而不愿意使用?这时候,一个简洁美观的图形用户界面(GUI)就派上用场了。 | ||
|
|
||
| 今天我们来介绍一个神奇的Python库——Gooey,它能让你的命令行程序秒变图形界面,完全不用写繁琐的GUI代码! | ||
|
|
||
| 10. [Cirrocumulus | 百万级单细胞数据集的可视化神器](https://mp.weixin.qq.com/s/3KxB7A_7_tF1I67fuHZWug) | ||
|
|
||
|  | ||
|
|
||
| Cirrocumulus可以读入多种单细胞、空间组学类型文件,交互式展示UMAP、dotplot、barplot等可视化结果。更重要的是,其不仅可以在本地运行,也可在云端展示数据,并通过分块读取单细胞数据,实现百万级单细胞数据集的秒加载。 | ||
|
|
||
| 11. [bcbio-nextgen | 基于Python的高效自动化生信分析工具](https://github.com/bcbio/bcbio-nextgen) | ||
|
|
||
| <img width="863" alt="image" src="https://github.com/user-attachments/assets/3a90b712-36d3-407d-a9aa-33b4f578f26d"> | ||
|
|
||
| bcbio-nextgen是一个社区驱动的生物信息学工具集,用于高通量的基因变异检测、RNA测序和小RNA分析。它通过一个配置文件实现自动化和并行处理,简化了从数据输入到分析的流程。该项目支持自动化验证和分布式计算,提供了安装脚本和文档,以促进研究人员专注于下游的生物学研究。 | ||
|
|
||
|
|
||
| ## 资源 | ||
|
|
||
| 12. [18个机器学习核心算法模型!!](https://mp.weixin.qq.com/s/q0j7O4CQPr20ygDO8Xi1bw) | ||
|
|
||
|
|
||
|  | ||
|
|
||
|
|
||
| 本篇推文中,作者介绍了18个机器学习核心算法模型,并提供了每种算法模型的核心公式和代码。 | ||
|
|
||
| 13. [Learn Next.js 中文教程](https://qufei1993.github.io/nextjs-learn-cn/) | ||
|
|
||
|  | ||
|
|
||
|
|
||
| 14. [comboSC](https://github.com/bm2-lab/comboSC) | ||
|
|
||
|  | ||
|
|
||
|
|
||
| comboSC是一个基于单细胞RNA-seq的个性化肿瘤联合治疗预测软件。它使用了大量来自CMAP和GDSC数据库的分子药物信息,可以基于癌症患者的单细胞测序数据,设计高效的免疫评分,用于个性化免疫谱评估。 | ||
|
|
||
| ## 历史上的本周 | ||
| - 2023 [生信爱好者周刊(第 89 期):视频学习胜过读书吗?](https://mp.weixin.qq.com/s/oGzJ4i9QxEM5GbBhNgsl6w) | ||
|
|
||
| ## 贡献者(GitHub ID) | ||
|
|
||
| 「Openbiox 生信周刊」运维小队: | ||
|
|
||
| - `@ShixiangWang`(王诗翔) | ||
| - `@kkjtmac`(阚科佳) | ||
| - `@NiEntropy`(赵启祥) | ||
| - `@He-Kai-fly`(何凯) | ||
| - `@JnanZhang`(张佳楠) | ||
| - `@Tomcxf`(陈啸枫) | ||
| - `@wangdepin`(王德品) | ||
| - `@kongjianyang`(空间阳) | ||
| - `@donghongyu2020`(董弘禹) | ||
| - `@DrRobinLuo`(罗鹏) | ||
|
|
||
| ## 订阅 | ||
|
|
||
| 这个周刊每周日发布,同步更新在微信公众号「优雅R」(elegant-r)上。 | ||
|
|
||
| 微信搜索“优雅R”或者扫描二维码,即可订阅。 | ||
|
|
||
|
|
||
|  | ||
|
|
||
|
|
||
| (完) |