2023-2 데이터사이언스 캡스톤디자인 8조
주제 : 기존 약물의 기타 질병치료로의 활용 가능성 확장(약물 재창출)
💡신약개발 연구는 많은 비용과 시간이 들지만 대부분 실패합니다. 여러 실험 데이터가 있는 기존 약물이 다른 질병 치료에 쓰일 수 있다면 매우 효율적일 것입니다. 절약된 자원은 환자의 비용 부담을 줄이고, 더 많은 의학 연구에 활용될 수 있습니다.

- 미디어커뮤니케이션학과 안혜린
- 생명과학과 김예원
- 생명과학과 이성경
- 생명과학과 전진아
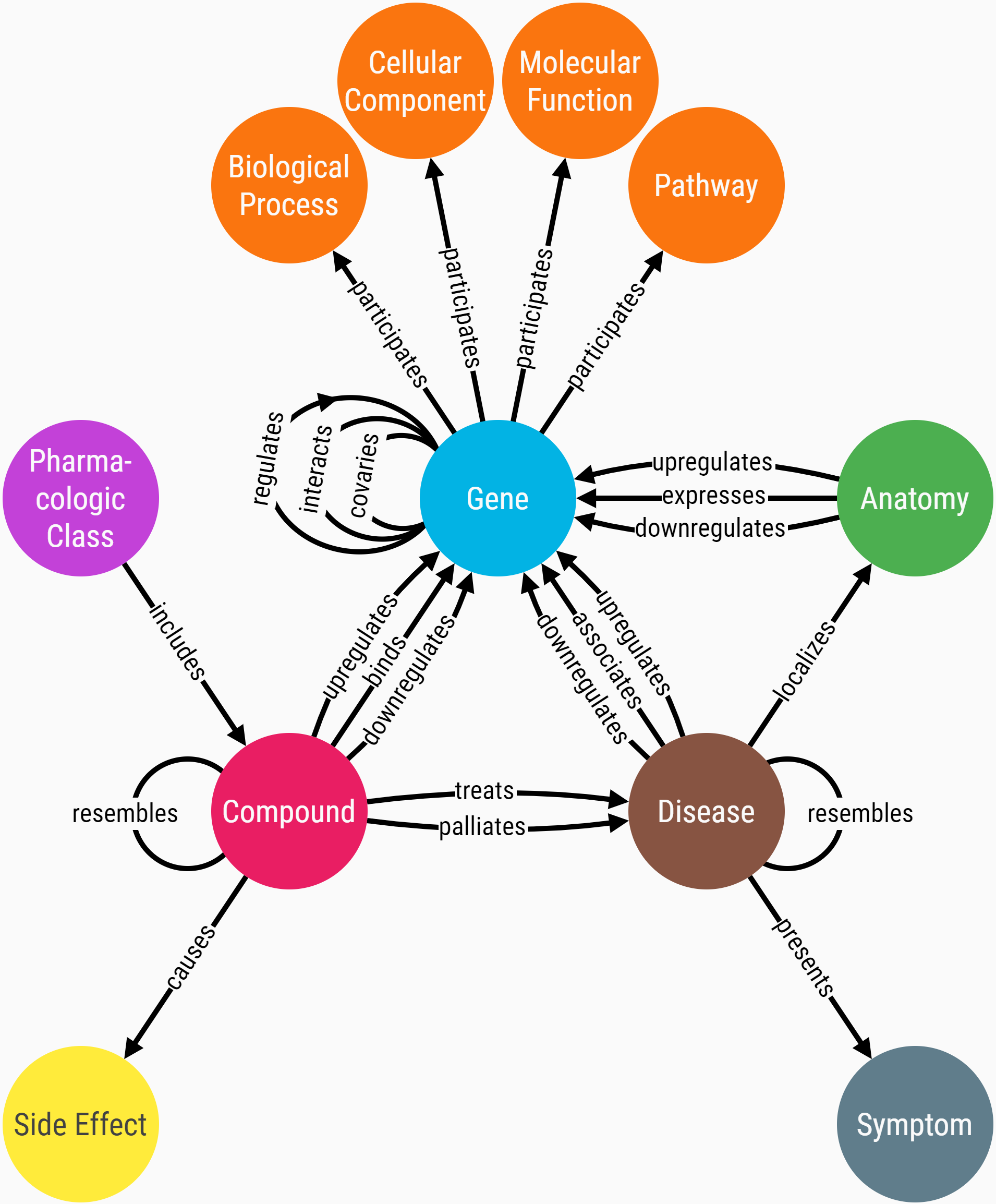
데이터 출처 : 생물 지식 그래프 오픈 네트워크 HETIONET
➡️이 중 compound, gene, disease의 세 노드와 서로 연관된 엣지만을 이용합니다.
유전자(gene)는 서로 유사한 진화적 변화를 겪었거나(covaries), 서로 상호작용하는 단백질을 생성하거나(interacts), 다른 유전자 발현에 관여(regulates)합니다.
생물학적으로 유사한 유전자는 유사한 기능을 공유합니다.
우리는 데이터 내 약물의 유전자와의 관계, 그 유전자의 관계들을 이용해
약물의 기존에 밝혀진 질병 치료뿐만 아니라 데이터 내 기타 질병 치료로의 활용 가능성을 확인하는 것을 목표로 합니다.
Bang, D., Lim, S., Lee, S., & Kim, S. (2023). Biomedical knowledge graph learning for drug repurposing by extending guilt-by-association to multiple layers. Nature Communications, 14, Article 3570. https://www.nature.com/articles/s41467-023-39301-y
Node2Vec은 그래프 데이터 내의 약물, 유전자, 질병의 관계를 이용해 임베딩할 수 있도록 하는 알고리즘입니다.
word2vec은 문장 안에서 단어 사이의 유사도를 이용해 임베딩한다면
node2vec은 그래프 안에서 다른 노드와의 관계를 이용해 노드를 저차원의 벡터 공간으로 투영시켜 임베딩합니다.
Grover, A., & Leskovec, J. (2016). node2vec: Scalable Feature Learning for Networks. arXiv preprint arXiv:1607.00653. https://arxiv.org/pdf/1607.00653
node2vec은 random walk로 그래프 내의 노드를 랜덤하게 이동해서 노드를 임베딩합니다.
우리는 파라미터를 조정해 완전 무작위 학습이 아닌, 필요한 정보를 이용해 노드 임베딩을 하는 것을 목표로 합니다.
임베딩된 각각의 약물, 질병 임베딩 값을 한 쌍으로 결합한 벡터를 input으로, 치료/완화 관계가 있는지 없는지의 binary classification을 진행합니다.
Label bias를 해결하기 위한 Random Over Sampling,
클래스별 예측 확률 신뢰도와 성능 개선을 위한 XGBoost를 선택했습니다.

가장 유의한 결과를 얻을 수 있는 breast cancer, 유방암 관련 예측 확률 상위10개의 후보약물 표입니다.
이미지를 눌러 pdf를 다운받으면, Evidences에 첨부된 선행연구자료를 확인할 수 있습니다.
2번을 제외하고 치료 효과에 대한 연구자료가 있으며, 6,8,9번의 경우 부작용에 대한 언급이 있어 검증이 필요합니다.